關於CCND1 promoter
由 EVKVLIN 在 二, 04/01/2014 – 14:52 發表 EBV and KSHV CTCF Rta senescence
03/25/2014
Dear 嬿如,
昨天張堯老師來電說第一次做出來的DAPA結果竟然與他們預測的不一樣 -> Rta binds to both the wild-type and the RRE mutated probes!所以我與他要了sequence來看看可能的解釋是什麼. (上次K156A的結果,有測CCND1嗎? Rta是直接結合到這個promoter上嗎? thank you)
———————
03/26/2014
Dear Dr. Lin,
上次K156A的結果在CCND1上也不是很明顯(EREV8中wild type : K156A= 0.7: 0.76, 而在ERKV中為0.87: 1.21) (如附件Fig.1)
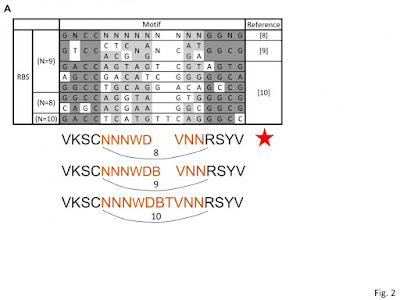
在CCND1 promoter上的potential RRE, 我是用5′-GNCC(N9)GGNG-3’預測出來的
在張堯老師所設計的probe片段中沒有其他位置有GNCC(N8-N10)GGNG或是CC(N9)GG等選擇
但是DAPA結果顯示Rta還是bind此段序列, 所以我猜core element的條件可能要更鬆一點
我試用另一個序列來預測RRE(如附件Fig. 2)
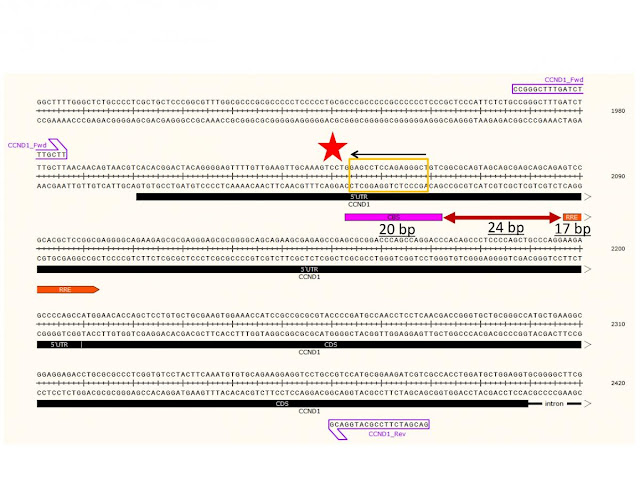
用interval region為8 bp的motif來搜尋probe片段, 則會發現CBS上可能有potential RBS! (如附件Fig.3的銘黃色方框)
您覺得呢?
#1由 sufang 在 一, 04/21/2014 – 11:41 發表。
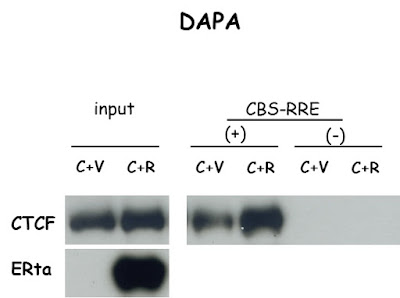
DAPA data
DAPA結果如附檔。(如先前討論過的,我們簡化實驗組別,拿掉了 RRE mutant control,以避免讓你們的故事主軸走岔了。)
在 Rta 存在的情形下,CTCF 對 CBS-RRE probe 的 in vitro binding 不會減弱 (甚至有些增強,已重覆兩次實驗)。
暗示在沒有 DNA methylation 的條件下,Rta 並不會抑制 CTCF 對 target site 的結合,這和你們 12~24h 的 ChIP 結果一致。
藉此可以導入你們提到的後期 Rta induces DNA methylation,因而讓 CTCF 離開 DNA。如有需要進一步討論的地方,請再與我聯絡。
#2由 EVKVLIN 在 二, 04/01/2014 – 15:03 發表。
希望不是
希望不是。第一,那你所有的CBS不是重新都要run一下有沒有這樣一種sequence. 第二,那凡是有這樣情形的CBS, Rta應該都會和CTCF競爭bind上去? MYC有嗎?
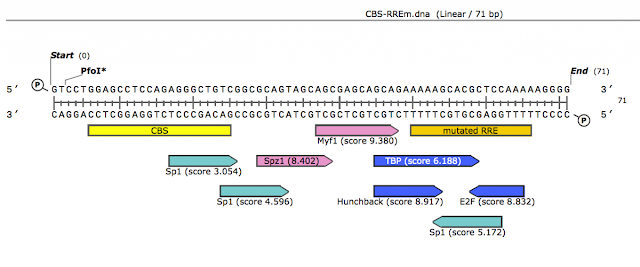
我Run了一下consite
還會有三個分數頗高的TF會binding上去的site (請見附檔的圖). 有沒有可能在RREm上再動些手腳?
4/2/2014 補上張堯老師給的LMP DAPA probe, 他們的就沒有在mutated probe上多出一個分數中等的Sp1 site, 而且還少掉一些TF binding site (吉).