#1由 ingrid 在 三, 01/14/2015 – 16:52 發表。
RT-qPCR protocol
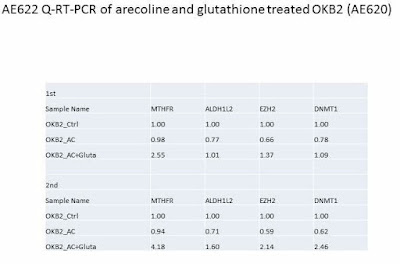
送去做RNA-seq的OKB2細胞的RNA,做了3次RT-qPCR,但結果似乎都不如預期,只選擇一次結果放上去。
#4由 sufang 在 六, 01/10/2015 – 20:32 發表。
Nature. 2015 Jan
Nature. 2015 Jan 8;517(7533):170-3. doi: 10.1038/nature14029.
PMID: 25567281
Reniere ML1, Whiteley AT2, Hamilton KL3, John SM1, Lauer P4, Brennan RG3, Portnoy DA5.
Author information
Abstract
Intracellular pathogens are responsible for much of the world-wide morbidity and mortality due to infectious diseases. To colonize their hosts successfully, pathogens must sense their environment and regulate virulence gene expression appropriately. Accordingly, on entry into mammalian cells, the facultative intracellular bacterial pathogen Listeria monocytogenes remodels its transcriptional program by activating the master virulence regulator PrfA. Here we show that bacterial and host-derived glutathione are required to activate PrfA. In this study a genetic selection led to the identification of a bacterial mutant in glutathione synthase that exhibited reduced virulence gene expression and was attenuated 150-fold in mice. Genome sequencing of suppressor mutants that arose spontaneously in vivo revealed a single nucleotide change in prfA that locks the protein in the active conformation (PrfA*) and completely bypassed the requirement for glutathione during infection. Biochemical and genetic studies support a model in which glutathione-dependent PrfA activation is mediated by allosteric binding of glutathione to PrfA. Whereas glutathione and other low-molecular-weight thiols have important roles in redox homeostasis in all forms of life, here we demonstrate that glutathione represents a critical signalling molecule that activates the virulence of an intracellular pathogen.
#5由 EVKVLIN 在 五, 12/12/2014 – 09:07 發表。
AE621 RNA-Seq analysis of AC-treated HOK cells_GA341
陳嬿如 <mmiiee313@gmail.com>
To: Tze-Tze Liu <ttliu@ym.edu.tw>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
To: Tze-Tze Liu <ttliu@ym.edu.tw>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
Dear 劉博士, 您好:
Mon, Nov 10, 2014 at 5:03 PM
我是國衛院癌研所林素芳老師的博士後研究員陳嬿如
在今年四月時, 有請您幫忙將計劃編號NSC101-2325-B002-059的經費核銷了8個ChIP-seq的核心使用費, 但 樣本⼀一直還未寄送給您
由於我畢業了, 研究方向也稍做改變, 因此想將這批經費改送8個mRNA-seq, 請問這樣經費的使用上是否足夠? 而最近是否方便送件呢?
若樣本濃度為200 ng/ul, total 10 ug 是否足夠呢?
另外, 寄件部分是否使用乾冰寄送就可以了? 親自送件的話, 最遲幾點送達您實驗室較好呢? 除此之外是否有其他需要注意的地方呢?
感謝您, 並請不吝指教!
由於我畢業了, 研究方向也稍做改變, 因此想將這批經費改送8個mRNA-seq, 請問這樣經費的使用上是否足夠? 而最近是否方便送件呢?
若樣本濃度為200 ng/ul, total 10 ug 是否足夠呢?
另外, 寄件部分是否使用乾冰寄送就可以了? 親自送件的話, 最遲幾點送達您實驗室較好呢? 除此之外是否有其他需要注意的地方呢?
感謝您, 並請不吝指教!
敬祝 研安 陳嬿如 敬上
037-246-166 ext 35122
Tze-Tze Liu <ttliu@ym.edu.tw>
To: 陳嬿如 <mmiiee313@gmail.com>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
To: 陳嬿如 <mmiiee313@gmail.com>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
於 2014/11/10 下午 05:03, 陳嬿如 提到: Dear 劉博士, 您好:
Fri, Nov 14, 2014 at 11:29 AM
我是國衛院癌研所林素芳老師的博士後研究員陳嬿如
在今年四月時, 有請您幫忙將計劃編號NSC101-2325-B002-059的經費核銷了8個 ChIP-seq的核心使用費, 但樣本⼀一直還未寄送給您
由於我畢業了, 研究方向也稍做改變, 因此想將這批經費改送8個mRNA-seq, 請問這樣經費的使用上是否足夠? 而最近是否方便送件呢?
在今年四月時, 有請您幫忙將計劃編號NSC101-2325-B002-059的經費核銷了8個 ChIP-seq的核心使用費, 但樣本⼀一直還未寄送給您
由於我畢業了, 研究方向也稍做改變, 因此想將這批經費改送8個mRNA-seq, 請問這樣經費的使用上是否足夠? 而最近是否方便送件呢?
早期 ChIP-seq 由於無法做 sample barcode,每個樣品要跑⼀一個 lane,核銷經 費有 628,000,這筆費用現在 可以做較多 ChIP 及 RNA 樣品的定序。
1. 如果做 8個 poly-A mRNA library preparation (NT$10,400*8=83,200), Illumina pair-end 2*100 bp sequencing 2lane(NT$94,500*2=189,000),合計NT$272,200,每個樣品的序列筆數約50millionreads 左右。
2. 如果做 8個 rRNA depleted RNA library preparation (NT$14,200*8=113,600), Illumina pair-end 2*100 bp sequencing 2lane(NT$94,500*2=189,000),合計NT$302,600,每個樣品的序列筆數約50millionreads左 右。⼀一般如果想看 non-coding RNA (miRNA 除外) 可以選擇用 rRNA depleted RNA sequencing,但是 seq read 可能會有 30-50% 是⼀一些 signal recognition particle RNA (eg. RN7SL1, RN7SL2 etc.),與 poly-A mRNA sequencing 相較需要較多的 reads 做分析。
若樣本濃度為200 ng/ul, total 10 ug 是否足夠呢?
RNA 最好以 DNase I 處理,避免 DNA 汙染干擾後面的資料分析。total RNA 200 ng/ul, total 10 ug 足夠,如
RNA 最好以 DNase I 處理,避免 DNA 汙染干擾後面的資料分析。total RNA 200 ng/ul, total 10 ug 足夠,如
1 of 3 12/12/14 9:02 AM
Gmail – 再次請教送件問題 https://mail.google.com/mail/u/0/?ui=2&ik=d85e4b4e1a&vi…
果樣品準備好就可以用乾冰寄來/送來,我們下午 6:30 前都有人在。如果採快遞寄件的話請避免星期五寄,以 免星期六無人收件。
抱歉! 延遲回覆。如有問題請與我聯絡,謝謝
孜孜
Tze-Tze Liu (劉孜孜)
Genome Research Center National Yang-Ming University Taipei, Taiwan 11221
Tel: 886-2-2826-7318 Fax: 886-2-2825-0305 e-mail: ttliu@ym.edu.tw
tze@pmf.tw
——————————
陳嬿如 <mmiiee313@gmail.com> Thu, Nov 27, 2014 at 3:47 PM To: Tze-Tze Liu <ttliu@ym.edu.tw>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
Dear 劉博士, 您好:
我們已將樣品準備好,於今日(11/27)下午用黑貓冷凍宅急便寄送給您,預計明天(11/28)中午前抵達,再請您 幫忙注意有沒有收到
這次送件有8個樣品,預定做 8個 rRNA depleted RNA library preparation (NT$14,200*8=113,600)和Illumina pair-end 2*100 bp sequencing 4 lane (NT$94,500*4=378,000),合計NT$491,600,這個計算方式是否正確? 在經費和實驗材料上的運用會不會造成您的不便?
這次送件有8個樣品,預定做 8個 rRNA depleted RNA library preparation (NT$14,200*8=113,600)和Illumina pair-end 2*100 bp sequencing 4 lane (NT$94,500*4=378,000),合計NT$491,600,這個計算方式是否正確? 在經費和實驗材料上的運用會不會造成您的不便?
非常感謝您通融我們能夠繼續運用這筆經費, 希望沒有造成您太大的困擾 若有任何問題再麻煩您與我連絡
實驗室電話: (037)246-166 分機35122
手機: 0958813027
實驗室電話: (037)246-166 分機35122
手機: 0958813027
敬祝 研安 陳嬿如 敬上
Dear 劉博士,
不好意思, 想再請教⼀一下, 若希望每個樣品都有100M reads, 所以⼀一個lane跑兩個樣本, 不知道這樣的算法是否 正確?
感謝您
嬿如 敬上
Tze-Tze Liu <ttliu@ym.edu.tw>
To: 陳嬿如 <mmiiee313@gmail.com>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
To: 陳嬿如 <mmiiee313@gmail.com>
Cc: Su-Fang Lin <sflin1@gmail.com>, 英潔 <ingridko@nhri.org.tw>
於 2014/11/27 下午 03:47, 陳嬿如 提到: Dear 劉博士, 您好:
Fri, Nov 28, 2014 at 7:08 PM
我們已將樣品 準備好,於今日(11/27)下午用黑貓冷凍宅急便寄送給您,預計明天(11/28)中午前抵達,再請 您幫忙注意有沒有收到
這次送件有8 個樣品,預定做 8個 rRNA depleted RNA library preparation (NT$14,200*8=113,600)和 Illumina pair-end 2*100 bp sequencing 4 lane (NT$94,500*4=378,000),合計NT$491,600,這個計算方式 是否正確?在經費和實驗材料上 的運用會不會造成您的不便?
這次送件有8 個樣品,預定做 8個 rRNA depleted RNA library preparation (NT$14,200*8=113,600)和 Illumina pair-end 2*100 bp sequencing 4 lane (NT$94,500*4=378,000),合計NT$491,600,這個計算方式 是否正確?在經費和實驗材料上 的運用會不會造成您的不便?
樣品已收到,我們下星期先做 RNA QC,如果 RNA 品質好的話就會排入實驗。費用的計算正確,2 個樣品跑 paired-end 會有 100-150 reads。
孜孜
Tze-Tze Liu AttachmentsDec 3 (9 days ago)
to 陳嬿如, me, 英潔
to 陳嬿如, me, 英潔
嬿如,
所送 8 RNA 樣品 QC 檢查如附件,所有樣品 RIN > 8,我們會排入實驗做 library。
孜孜
#6由 sufang 在 四, 10/16/2014 – 11:11 發表。
Epigenetics 2012: Supra-physiological folic acid concentrations
這一篇說4.5–68 nM為生理濃度,大於1 uM則為超生理濃度。一般medium中似乎為超生理? http://www.ncbi.nlm.nih.gov/pubmed/22617627
#7由 sufang 在 三, 10/15/2014 – 15:58 發表。
medium中folic acid濃度
OFK4/OKB2/OKF4-hTert:
1/4 DMEM (4 mg/L) + 1/4 F12 (1.3 mg/L) + 1/2 KSFM (待查) = 3 uM
Stromal/Fibroblast-htert:
1/2 M106 (待查) + 1/2 M199 (0.01 mg/L) = 10 nM
#8由 ingrid 在 三, 10/15/2014 – 14:53 發表。
20141007 lab meeting
#9由 sufang 在 三, 12/24/2014 – 14:36 發表。
原HOK相關實驗post and comment
7/17/2014 SFL:
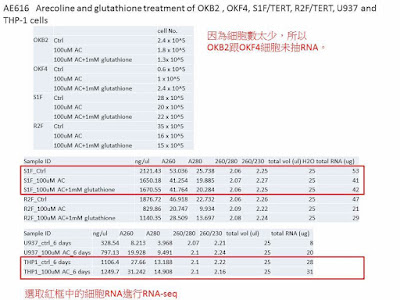
1. 二、三月間買來、名為OKB2, OKB8, OKF4的HOK,皆為p4。 目前已成功解出二株。細胞提供人Rheinwald Lab建議,HOK不要養超過1/3滿,或是不要超過8天,而且繼代用的medium與作實驗用的medium稍稍不同。我們家遵照他們的指示,繼代時均 不養超過1/3滿,等到要treatment的前一天,換成作實驗用的medium,這樣子細胞可以較confluent,收下來的蛋白質或RNA也都還 夠做一般下游實驗使用。
2. 另外也買了兩株hTert-immortalized fibroblast. 目前還沒時間有解凍、分裝。
3. 因為考慮到fibroblast及macrophage可能產生的加成反應,想一律用transwell系統,把HOK擺下層,上面放fibroblast/macrophage。另一方面,RNA-SEQ需要RIN>8的好RNA 5-ug.
4. HOK平均doublining time約為40-50小時,決定所有東西處理5天。
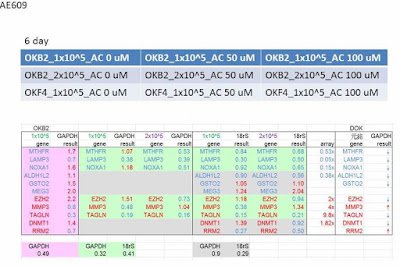
5. Arecoline (AC)濃度: 50 uM–100uM。
9/19/2014 (Fri) Ingrid post 初步結果
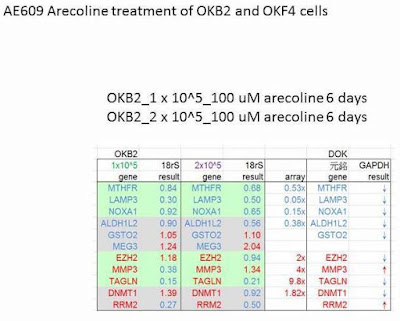
1 x 10^5 OKB2用100uM arecoline處理6天收的RNA,轉RT後做Q-PCR的結果如下